Inibitore irreversibile
Gli inibitori irreversibili sono inibitori enzimatici che si legano covalentemente o che distruggono un gruppo funzionale dell'enzima essenziale per la sua attività.
Tipi[modifica | modifica wikitesto]

Gli inibitori irreversibili solitamente modificano covalentemente l'enzima, e l'inibizione non può essere pertanto successivamente neutralizzata. Spesso tali inibitori contengono gruppi funzionali reattivi come aldeidi, aloalcani o alcheni. Questi gruppi elettrofili reagiscono con le catene laterali degli amminoacidi formando addotti covalenti. I residui modificati sono quelli con catene laterali contenenti gruppi nucleofili come l'ossidrile o il sulfidrile (ad esempio la serina, bersagliabile dal diisopropilfluorofosfato, la cisteina, la treonina o la tirosina).[1]
Il concetto di inibizione irreversibile è in ogni caso diverso da quello di inattivazione irreversibile dell'enzima. Gli inibitori irreversibili sono solitamente specifici per una classe di enzimi e non inattivano tutte le proteine; non funzionano distruggendo la struttura proteica ma alterando specificamente il sito attivo del loro bersaglio. Gli inibitori irreversibili, dunque, non sono correlati a fenomeni legati a livelli estremi di pH o di temperatura, che causano solitamente la denaturazione aspecifica di tutte le strutture proteiche presenti in soluzione, oppure all'idrolisi aspecifica mediata dall'aggiunta nel mezzo di acido cloridrico concentrato caldo, che genera una rottura dei legami peptidici ed un rilascio di amminoacidi liberi.[2]
Analisi dell'inibizione irreversibile[modifica | modifica wikitesto]
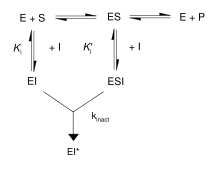
Gli inibitori irreversibili formano un complesso reversibile non-covalente con l'enzima (EI o ESI), che reagisce per produrre un complesso covalentemente modificato e definito EI* o dead-end (senza uscita). Il rapporto a cui si forma EI* è detto rapporto di inattivazione (o kinact). Dal momento che la formazione di EI può competere con ES, il legame degli inibitori irreversibili può essere evitato mediante competizione con un substrato o con un secondo inibitore reversibile.
I meccanismi di legame e di inattivazione di questo tipo di reazione possono essere investigati incubando l'enzima e l'inibitore e misurando la quantità di attività rimanente al trascorrere del tempo. L'attività decresce in maniera dipendente dal tempo, di solito seguendo il decadimento esponenziale. Riportando questi dati in una equazione di rapporto si ottiene il tasso di inattivazione a quella concentrazione dell'inibitore. Questa operazione è ripetuta a diverse concentrazioni dell'inibitore. Se un complesso reversibile EI è coinvolto, il tasso di inattivazione sarà saturabile e, disegnando la curva, si otterrà la kinact e la Ki.[3]
Un altro metodo largamente usato in queste analisi è la spettrometria di massa. In questo caso un'accurata misura della massa dell'enzima originario non modificato e dell'enzima inattivato permette di calcolare la crescita in massa causata dalla reazione con l'inibitore e mostra la stechiometria della reazione. Questo è indagabile usando uno spettrometro di massa MALDI-TOF. Una tecnica complementare, l'impronta digitale proteica, implica la digestione della proteina originaria e modificata mediante una proteasi come la tripsina. Ciò produce un insieme di peptidi che possono essere analizzati con uno spettrometro di massa. Il peptide la cui massa cambia dopo la reazione con l'inibitore è quello che contiene il sito di modificazione.
Casi particolari[modifica | modifica wikitesto]

Spesso alcuni inibitori reversibili sono in grado di legarsi così strettamente al loro bersaglio da apparire come irreversibili. Questi inibitori mostrano costanti cinetiche simili a quelle degli inibitori irreversibili covalenti. In questi casi, alcuni di questi inibitori si legano rapidamente all'enzima nel complesso a bassa affinità EI e questo successivamente va incontro ad un più lento riassestamento verso un complesso EI* legato molto saldamente. Tale comportamento cinetico è definito legame lento[5] Questo lento assestamento dopo il legame provoca spesso una modifica di conformazione, legata al fatto che l'enzima si stringe attorno alla molecola dell'inibitore. Esempi di inibitori a legame lento sono costituiti da importanti farmaci, come il metotrexato[6], l'allopurinolo[7] e la forma attivata dell'aciclovir.[8]
Esempi di inibitori irreversibili[modifica | modifica wikitesto]

Il diisopropilfluorofosfato (DFP) è un inibitore delle proteasi, che idrolizza il legame fosforo-fluoro lasciando il fosfato legato alla serina presente nel sito attivo, inibendo in modo irreversibile l'enzima.[9] In modo analogo, il DFP reagisce anche col sito attivo dell'acetilcolinesterasi nelle sinapsi dei neuroni, ed è quindi anche una potente neurotossina, la cui dose letale è fissata a meno di 100 mg.[10]
Un altro esempio di inibitore irreversibile delle proteasi è la iodoacetamide, che si lega ad un residuo di cisteina dell'enzima. Lo zolfo contenuto nell'aminoacido reagisce con tale inibitore, si forma un tioetere liberando acido ioidrico (HI). L'enzima quindi perde la sua attività.
L'inibizione suicida è un tipo inusuale di inibizione irreversibile che si ha quando l'enzima trasforma l'inibitore in una forma reattiva nel suo sito attivo. Un esempio è l'α-difluorometilornitina (o DFMO), inibitore della biosintesi delle poliammine ed analogo dell'amminoacido ornitina, usato per trattare la tripanosomiasi africana (febbre del sonno). L'ornitina decarbossilasi può catalizzare la decarbossilazione del DFMO invece che dell'ornitina. Conseguenza di tale reazione è l'eliminazione di un atomo di fluoro, che converte questo intermedio catalitico nell'immina coniugata, una specie altamente elettrofila, che reagisce con un residuo di cisteina o di lisina nel sito attivo per inibire irreversibilmente l'enzima.[4]
Dal momento che l'inibizione irreversibile dà spesso luogo all'iniziale formazione di un complesso non-covalente EI, è talvolta possibile per un inibitore legarsi ad un enzima in più di un modo. Per esempio, un inibitore chiamato mostarda di chinacrina può inibire in due modi diversi la tripanotione reduttasi del protozoo Trypanosoma cruzi. In un caso può legarsi reversibilmente, nell'altro covalentemente, dal momento che reagisce con un residuo amminoacidico attraverso la sua azoiprite.[11]
Inibitori suicidi[modifica | modifica wikitesto]
Particolare classe di inibitori irreversibili.
Note[modifica | modifica wikitesto]
- ^ Lundblad R. L. Chemical Reagents for Protein Modification CRC Press Inc (2004) ISBN 0-8493-1983-8
- ^ N. Price, B. Hames, D. Rickwood (Ed.) Proteins LabFax Academic Press (1996) ISBN 0-12-564710-7
- ^ Maurer T, Fung HL. Comparison of Methods for Analyzing Kinetic Data From Mechanism-Based Enzyme Inactivation: Application to Nitric Oxide Synthase. AAPS PharmSci. (2000) 2(1)E8. PMID 11741224
- ^ a b Poulin R, Lu L, Ackermann B, Bey P, Pegg AE. Mechanism of the irreversible inactivation of mouse ornithine decarboxylase by alpha-difluoromethylornithine. Characterization of sequences at the inhibitor and coenzyme binding sites. Archiviato il 24 gennaio 2009 in Internet Archive. J Biol Chem. 1992 Jan 5;267(1):150–8. PMID 1730582
- ^ Szedlacsek, S.E. and Duggleby, R.G. Kinetics of slow and tight-binding inhibitors. Meth. Enzymol., (1995) 249: 144–180. PMID 7791610
- ^ Stone SR, Morrison JF. Mechanism of inhibition of dihydrofolate reductases from bacterial and vertebrate sources by various classes of folate analogues. Biochim Biophys Acta. 1986 Feb 14;869(3):275–85. PMID 3511964
- ^ Hille R, Massey V. Tight binding inhibitors of xanthine oxidase. Pharmacol Ther. 1981;14(2):249–63. PMID 4322209
- ^ Reardon JE. Herpes simplex virus type 1 and human DNA polymerase interactions with 2'-deoxyguanosine 5'-triphosphate analogues. Kinetics of incorporation into DNA and induction of inhibition. Archiviato il 24 gennaio 2009 in Internet Archive. J Biol Chem. 1989 Nov 15;264(32):19039–44. PMID 2553730
- ^ J. A. Cohen , R. A. Oosterbaan and F. Berends Organophosphorus compounds Meth. Enzymol. (1967) 11, 686
- ^ Brenner, G. M. (2000): Pharmacology. Philadelphia, PA: W.B. Saunders Company. ISBN 0-7216-7757-6
- ^ Saravanamuthu A, Vickers TJ, Bond CS, Peterson MR, Hunter WN, Fairlamb AH. Two interacting binding sites for quinacrine derivatives in the active site of trypanothione reductase: a template for drug design. Archiviato il 12 maggio 2009 in Internet Archive. J Biol Chem. 2004 Jul 9;279(28):29493–500. PMID 15102853
Bibliografia[modifica | modifica wikitesto]
- David L. Nelson, Michael M. Cox, I Principi di Biochimica di Lehninger, 3ª ed., Bologna, Zanichelli, febbraio 2002, ISBN 88-08-09035-3.
Voci correlate[modifica | modifica wikitesto]
- Inibitore enzimatico
- Inibitore reversibile
- Inibitore competitivo
- Inibitore non competitivo
- Inibitore acompetitivo
- Inibitore di tipo misto
- Inibizione enzimatica retroattiva da prodotto finale
- Inibitore suicida
- Enzima
- Catalisi enzimatica
- Regolazione allosterica
- Sito attivo
- Cinetica di Michaelis-Menten
- Costante di Michaelis-Menten
Collegamenti esterni[modifica | modifica wikitesto]
- (EN) Web tutorial on enzyme inhibition, dispensa di Peter Birch della University of Paisley, con animazioni
- (EN) Symbolism and Terminology in Enzyme Kinetics, consigli sulla terminologia di inibizione enzimatica ad opera del Nomenclature Committee of the International Union of Biochemistry (NC-IUB)
- (EN) PubChem from NCBI, database di farmaci ed inibitori enzimatici
- (EN) BRENDA Archiviato l'11 dicembre 2008 in Internet Archive., database degli enzimi con i relativi inibitori noti
- (EN) Enzymes, Kinetics and Diagnostic Use Archiviato il 9 luglio 2011 in Internet Archive., testo sulle applicazioni mediche degli inibitori enzimatici di Michael W. King della IU School of Medicine